栏目:生命科学 作者:佚名
黏连蛋白Cohesin介导细胞分裂过程中姐妹染色体的黏连,还参与染色质三维结构的形成和维持1,对哺乳动物细胞基因组DNA的稳定性有着非常重要的影响。黏连蛋白的失活突变在多种癌症中高频出现,但前期的研究表明黏连蛋白功能缺失导致的姐妹染色体黏连错乱引起的染色体非整倍性可能不是致癌的主要原因2,这表明黏连蛋白功能缺失导致基因组不稳定乃至肿瘤发生的分子机制仍待进一步挖掘。
2023年7月27日,北京大学生命科学学院和北大-清华生命科学联合中心胡家志课题组在Nature Genetics发文揭示了黏连蛋白Cohesin功能缺失导致致癌基因突变的分子机制。他们发现黏连蛋白缺失会导致休眠复制源在S期早期的大量激活,从而干扰正常的DNA复制时序,进而导致基因组不稳定性的剧增,从而引起部分致癌或者抑癌基因的突变。该研究是本课题组转录调节DNA复制起始的工作的延续,也拓展了他们对基因组三维结构调节DNA复制而维持基因组稳定性的分子机制的理解,还纠正了领域内早前部分研究认为黏连蛋白失活不影响DNA复制时序的错误认识。
为探究黏连蛋白失活的影响,作者在人K562免疫细胞和小鼠胚胎干细胞中构建了生长素介导的Degron系统,用以快速降解黏连蛋白Cohesin的核心组分之一——RAD21(图1a)。与之前报道一致,该系统只能部分降解RAD21(~70%)。为排除RAD21未降解细胞对实验结果的干扰,作者仅分选RAD21彻底降解的细胞用于后续分析(图1b),而之前的两项相关研究均未排除这部分细胞的影响,因此对实验结果造成了干扰。作者首先采用实验室前期开发的检测DNA双链断裂的高通量测序方法——PEM-seq3进行定量分析(Cell Discovery | 胡家志研究组开发出优化基因编辑和追踪DNA修复的新方法),发现RAD21缺失将导致全基因组的DNA断裂水平提高至原来的3-5倍(图1c)。这说明黏连蛋白在维持基因组稳定性方面发挥着极为重要的作用。
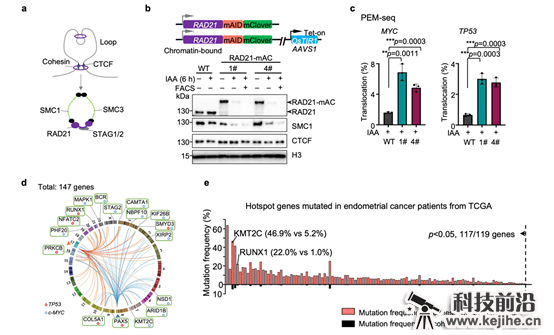
图1 RAD21降解造成大量基因组双链断裂发生。
(a)染色质loop及 Cohesin复合体示意图。(b)K562细胞中RAD21 AID蛋白降解体系构建与验证。1#和4#为构建成功的RAD21-mAID-mClover细胞系,简称RAD21-mAC。(c)PEM-seq方法从MYC和TP53位点分别检测RAD21降解后染色体易位频率。(d)RAD21降解导致的DSB热点基因。(e)TCGA数据库子宫内膜癌样本中热点基因与Cohesin蛋白亚基共突变情况。
进一步分析发现RAD21缺失导致的DNA断裂富集于147个热点基因,其中超过三分之一与癌症和其它疾病高度相关(图1d)。此外,这些热点损伤基因与黏连蛋白的失活存在显著共突变的关系(图1e),这表明黏连蛋白的失活突变可能是相关癌症或疾病发生发展的核心原因之一。通过分析这些DNA损伤的分布位置发现,染色质三维结构的改变和转录水平的轻微扰动不是黏连蛋白降解导致DNA损伤的主要原因。但作者发现RAD21降解导致的DNA损伤非对称性地分布于DNA双链,且该非对称分布与DNA复制过程中后随链的关键产物——冈崎片段的分布方向一致(图2a)。此外,黏连蛋白的降解还导致DNA复制速率下降且停顿复制叉增多(图2b, c),这些数据均表明黏连蛋白与DNA复制的稳定有着重要的关系,其缺失可能通过影响DNA复制而导致基因组不稳定性。目前已知DNA复制是细胞基因组不稳定性最大的来源。作者通过绘制细胞的DNA复制时序,发现黏连蛋白的降解导致了约30%基因组区域的DNA复制时序提前(图2d, e),且DNA损伤的热点基因大多位于DNA复制时序异常的区域(图2f)。
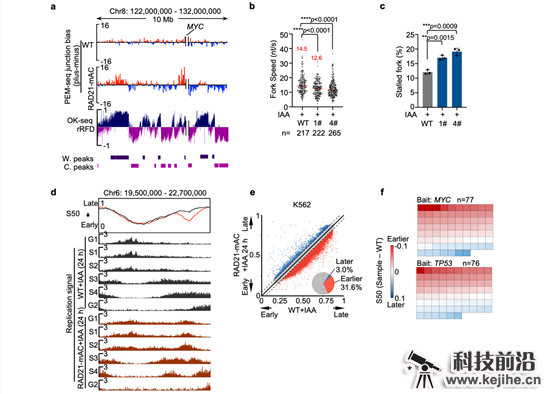
图2 RAD21降解导致复制时序紊乱和复制压力。
(a)PEM-seq检测RAD21降解后细胞中染色体易位断裂位点偏向性与OK-seq相似。(b)RAD21降解导致复制叉速率减慢。(c)RAD21降解导致复制叉停顿增加。(d)RAD21降解导致部分基因组区域复制时序提前。(e)K562细胞基因组中31.6%的区域在RAD21降解后发生复制时序的显著提前。(f)热点基因的复制时序变化情况,每个方格表示一个基因,红色和蓝色分别表示复制时序的提早和延迟。
为探究复制时序改变的原因,作者采用了该实验室前期开发的用于鉴定DNA复制起始的测序方法——NAIL-seq4(Genome Biology | 胡家志实验室揭示转录调节DNA复制起始的分子机制),发现RAD21的降解导致约25%更多的DNA复制源在S期早期被激活(图3a, b)。这些早期复制起始位点具有经典复制源的特征,为休眠复制源(dormant origins)。这些在早期被异常激活的复制源导致了DNA复制时序的提前(图3c)。休眠复制源在正常情况下只在DNA复制中晚期偶尔起始,只有在面临DNA复制压力时才会大量激活。而休眠复制源在S期早期的提前激活需要消耗更多的复制相关因子,从而引起复制压力,这可能是RAD21缺失导致基因组不稳定性的重要原因之一。定点敲除c-MYC基因附近的染色质环的锚点,将导致染色质环内部作用的减弱,从而引起DNA复制起始在染色质环内部的激活,并进而引起DNA损伤水平的增加(图3d)。这些数据揭示了Cohesin缺失→DNA复制起始位点增加→DNA复制时序紊乱→DNA损伤水平上升的逻辑关系,为深入理解黏连蛋白Cohesin在DNA复制过程中的功能提供了更多的解释。
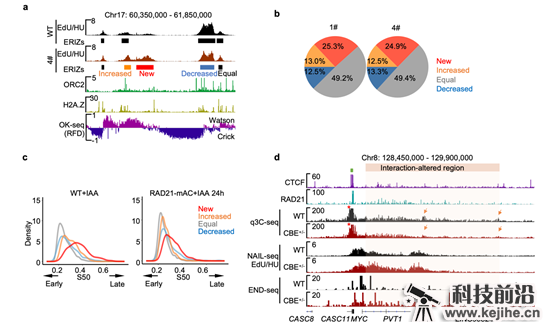 图3 RAD21降解导致复制起始位点增加并引起DNA双链断裂。
图3 RAD21降解导致复制起始位点增加并引起DNA双链断裂。
(a)RAD21降解后(4#)NAIL-seq检测到的早期复制起始信号(EdU/HU)和所鉴定的早期复制起始位点(Early replication initiation zones, ERIZs)。 (b)饼图统计1#和4#RAD21-mAC细胞中RAD21降解后ERIZ信号强度分类和比例。(c)K562中四类ERIZ区域在RAD21未降解和降解的细胞中所处的复制时序密度图。(d)c-MYC区域染色质环锚定位点的敲除(绿色方块)造成橙色阴影区域染色质相互作用(q3C-seq)减弱,早期复制起始信号(NAIL-seq)增强和DSB(END-seq)的增加。
生命科学学院、北大清华生命科学联合中心胡家志研究员为通讯作者。前沿交叉学院2023届博士毕业生吴锦淳、生命科学学院博士后刘阳博士、生命科学学院2021级博士研究生张丁峥嵘和刘栩豪为共同第一作者。艾晨博士、甘婷婷博士、梁昊昕、郭岳峰、陈莫晗、尹健行博士、刘懿阳和张微微博士亦有贡献。该工作得到了科技部、国家自然科学基金、细胞增殖与分化教育部重点实验室、北大-清华生命科学联合中心、生命科学学院、生命科学学院仪器平台以及凤凰中心的大力支持。
原文链接:https://rdcu.be/dhUmX
参考文献:
1. Rao, S.S.P., Huang, S.-C., Glenn St Hilaire, B., Engreitz, J.M., Perez, E.M., Kieffer-Kwon, K.-R., Sanborn, A.L., Johnstone, S.E., Bascom, G.D., Bochkov, I.D., et al. (2017). Cohesin Loss Eliminates All Loop Domains. Cell 171, 305-320.e324.
2. Waldman, T. (2020). Emerging themes in cohesin cancer biology. Nature Reviews Cancer 20, 504-515.
3. Yin, J., Liu, M., Liu, Y., Wu, J., Gan, T., Zhang, W., Li, Y., Zhou, Y., and Hu, J. (2019). Optimizing genome editing strategy by primer-extension-mediated sequencing. Cell discovery 5, 1-11.
4. Liu, Y., Ai, C., Gan, T., Wu, J., Jiang, Y., Liu, X., Lu, R., Gao, N., Li, Q., and Ji, X. (2021). Transcription shapes DNA replication initiation to preserve genome integrity. Genome biology 22, 1-27.
2023年7月27日,北京大学生命科学学院和北大-清华生命科学联合中心胡家志课题组在Nature Genetics发文揭示了黏连蛋白Cohesin功能缺失导致致癌基因突变的分子机制。他们发现黏连蛋白缺失会导致休眠复制源在S期早期的大量激活,从而干扰正常的DNA复制时序,进而导致基因组不稳定性的剧增,从而引起部分致癌或者抑癌基因的突变。该研究是本课题组转录调节DNA复制起始的工作的延续,也拓展了他们对基因组三维结构调节DNA复制而维持基因组稳定性的分子机制的理解,还纠正了领域内早前部分研究认为黏连蛋白失活不影响DNA复制时序的错误认识。
为探究黏连蛋白失活的影响,作者在人K562免疫细胞和小鼠胚胎干细胞中构建了生长素介导的Degron系统,用以快速降解黏连蛋白Cohesin的核心组分之一——RAD21(图1a)。与之前报道一致,该系统只能部分降解RAD21(~70%)。为排除RAD21未降解细胞对实验结果的干扰,作者仅分选RAD21彻底降解的细胞用于后续分析(图1b),而之前的两项相关研究均未排除这部分细胞的影响,因此对实验结果造成了干扰。作者首先采用实验室前期开发的检测DNA双链断裂的高通量测序方法——PEM-seq3进行定量分析(Cell Discovery | 胡家志研究组开发出优化基因编辑和追踪DNA修复的新方法),发现RAD21缺失将导致全基因组的DNA断裂水平提高至原来的3-5倍(图1c)。这说明黏连蛋白在维持基因组稳定性方面发挥着极为重要的作用。

图1 RAD21降解造成大量基因组双链断裂发生。
(a)染色质loop及 Cohesin复合体示意图。(b)K562细胞中RAD21 AID蛋白降解体系构建与验证。1#和4#为构建成功的RAD21-mAID-mClover细胞系,简称RAD21-mAC。(c)PEM-seq方法从MYC和TP53位点分别检测RAD21降解后染色体易位频率。(d)RAD21降解导致的DSB热点基因。(e)TCGA数据库子宫内膜癌样本中热点基因与Cohesin蛋白亚基共突变情况。
进一步分析发现RAD21缺失导致的DNA断裂富集于147个热点基因,其中超过三分之一与癌症和其它疾病高度相关(图1d)。此外,这些热点损伤基因与黏连蛋白的失活存在显著共突变的关系(图1e),这表明黏连蛋白的失活突变可能是相关癌症或疾病发生发展的核心原因之一。通过分析这些DNA损伤的分布位置发现,染色质三维结构的改变和转录水平的轻微扰动不是黏连蛋白降解导致DNA损伤的主要原因。但作者发现RAD21降解导致的DNA损伤非对称性地分布于DNA双链,且该非对称分布与DNA复制过程中后随链的关键产物——冈崎片段的分布方向一致(图2a)。此外,黏连蛋白的降解还导致DNA复制速率下降且停顿复制叉增多(图2b, c),这些数据均表明黏连蛋白与DNA复制的稳定有着重要的关系,其缺失可能通过影响DNA复制而导致基因组不稳定性。目前已知DNA复制是细胞基因组不稳定性最大的来源。作者通过绘制细胞的DNA复制时序,发现黏连蛋白的降解导致了约30%基因组区域的DNA复制时序提前(图2d, e),且DNA损伤的热点基因大多位于DNA复制时序异常的区域(图2f)。

图2 RAD21降解导致复制时序紊乱和复制压力。
(a)PEM-seq检测RAD21降解后细胞中染色体易位断裂位点偏向性与OK-seq相似。(b)RAD21降解导致复制叉速率减慢。(c)RAD21降解导致复制叉停顿增加。(d)RAD21降解导致部分基因组区域复制时序提前。(e)K562细胞基因组中31.6%的区域在RAD21降解后发生复制时序的显著提前。(f)热点基因的复制时序变化情况,每个方格表示一个基因,红色和蓝色分别表示复制时序的提早和延迟。
为探究复制时序改变的原因,作者采用了该实验室前期开发的用于鉴定DNA复制起始的测序方法——NAIL-seq4(Genome Biology | 胡家志实验室揭示转录调节DNA复制起始的分子机制),发现RAD21的降解导致约25%更多的DNA复制源在S期早期被激活(图3a, b)。这些早期复制起始位点具有经典复制源的特征,为休眠复制源(dormant origins)。这些在早期被异常激活的复制源导致了DNA复制时序的提前(图3c)。休眠复制源在正常情况下只在DNA复制中晚期偶尔起始,只有在面临DNA复制压力时才会大量激活。而休眠复制源在S期早期的提前激活需要消耗更多的复制相关因子,从而引起复制压力,这可能是RAD21缺失导致基因组不稳定性的重要原因之一。定点敲除c-MYC基因附近的染色质环的锚点,将导致染色质环内部作用的减弱,从而引起DNA复制起始在染色质环内部的激活,并进而引起DNA损伤水平的增加(图3d)。这些数据揭示了Cohesin缺失→DNA复制起始位点增加→DNA复制时序紊乱→DNA损伤水平上升的逻辑关系,为深入理解黏连蛋白Cohesin在DNA复制过程中的功能提供了更多的解释。

(a)RAD21降解后(4#)NAIL-seq检测到的早期复制起始信号(EdU/HU)和所鉴定的早期复制起始位点(Early replication initiation zones, ERIZs)。 (b)饼图统计1#和4#RAD21-mAC细胞中RAD21降解后ERIZ信号强度分类和比例。(c)K562中四类ERIZ区域在RAD21未降解和降解的细胞中所处的复制时序密度图。(d)c-MYC区域染色质环锚定位点的敲除(绿色方块)造成橙色阴影区域染色质相互作用(q3C-seq)减弱,早期复制起始信号(NAIL-seq)增强和DSB(END-seq)的增加。
生命科学学院、北大清华生命科学联合中心胡家志研究员为通讯作者。前沿交叉学院2023届博士毕业生吴锦淳、生命科学学院博士后刘阳博士、生命科学学院2021级博士研究生张丁峥嵘和刘栩豪为共同第一作者。艾晨博士、甘婷婷博士、梁昊昕、郭岳峰、陈莫晗、尹健行博士、刘懿阳和张微微博士亦有贡献。该工作得到了科技部、国家自然科学基金、细胞增殖与分化教育部重点实验室、北大-清华生命科学联合中心、生命科学学院、生命科学学院仪器平台以及凤凰中心的大力支持。
原文链接:https://rdcu.be/dhUmX
参考文献:
1. Rao, S.S.P., Huang, S.-C., Glenn St Hilaire, B., Engreitz, J.M., Perez, E.M., Kieffer-Kwon, K.-R., Sanborn, A.L., Johnstone, S.E., Bascom, G.D., Bochkov, I.D., et al. (2017). Cohesin Loss Eliminates All Loop Domains. Cell 171, 305-320.e324.
2. Waldman, T. (2020). Emerging themes in cohesin cancer biology. Nature Reviews Cancer 20, 504-515.
3. Yin, J., Liu, M., Liu, Y., Wu, J., Gan, T., Zhang, W., Li, Y., Zhou, Y., and Hu, J. (2019). Optimizing genome editing strategy by primer-extension-mediated sequencing. Cell discovery 5, 1-11.
4. Liu, Y., Ai, C., Gan, T., Wu, J., Jiang, Y., Liu, X., Lu, R., Gao, N., Li, Q., and Ji, X. (2021). Transcription shapes DNA replication initiation to preserve genome integrity. Genome biology 22, 1-27.
版权声明:文章观点仅代表作者观点,作为参考,不代表本站观点。部分文章来源于网络,如果网站中图片和文字侵犯了您的版权,请联系我们及时删除处理!转载本站内容,请注明转载网址、作者和出处,避免无谓的侵权纠纷。
相关推荐

